要旨 ディープネットワークの学習を成功させるには、数千もの注釈付き学習サンプルが必要であることは広く認められています。本稿では、データ拡張を効果的に活用することで、利用可能な注釈付きサンプルをより効率的に使用するネットワークと学習戦略を紹介します。このアーキテクチャは、コンテキストを捉える収縮パスと、正確な位置特定を可能にする対称拡張パスで構成されています。このようなネットワークは、非常に少ない画像からエンドツーエンドで学習でき、電子顕微鏡スタック内の神経構造のセグメンテーションに関するISBIチャレンジにおいて、これまでの最良の手法(スライディングウィンドウ畳み込みネットワーク)よりも優れた性能を発揮することを示します。透過光顕微鏡画像(位相差およびDIC)で学習した同じネットワークを使用することで、これらのカテゴリでISBI細胞追跡チャレンジ2015を大差で優勝しました。さらに、このネットワークは高速です。最新のGPUを使用すれば、512x512画像のセグメンテーションは1秒未満で完了します。完全な実装(Caeベース)と学習済みネットワークは、http://lmb.informatik.uni-freiburg.de/people/ronneber/u-net で入手できます。
過去2年間で、深層畳み込みネットワークは多くの視覚認識タスクにおいて最先端の性能を上回るようになりました(例:[7,3])。畳み込みネットワークは以前から存在していましたが([8])、利用可能な学習セットのサイズと対象となるネットワークのサイズのために、その成功は限られていました。Krizhevskyらによるブレークスルー([7])は、100万枚の学習画像を含むImageNetデータセットを用いて、8層、数百万のパラメータを持つ大規模なネットワークを教師あり学習したことによるものです。それ以来、さらに大規模で深いネットワークが学習されてきました([12])。
畳み込みネットワークの典型的な用途は分類タスクであり、画像への出力は単一のクラスラベルです。しかし、多くの視覚タスク、特に生物医学画像処理では、望ましい出力には位置特定、つまり各ピクセルにクラスラベルが割り当てられていることが必要です。さらに、生物医学タスクで数千枚のトレーニング画像を用意することは通常不可能です。そこで、Ciresanら[1]は、スライディングウィンドウ設定でネットワークをトレーニングし、各ピクセルの周囲の局所領域(パッチ)を入力として与えて、そのピクセルのクラスラベルを予測しました。まず、このネットワークは位置特定が可能です。次に、パッチで表されるトレーニングデータは、トレーニング画像の数よりもはるかに大きいです。この結果得られたネットワークは、ISBI 2012のEMセグメンテーションチャレンジで大差で優勝しました。
Ciresanら[1]の戦略には、明らかに2つの欠点があります。第一に、ネットワークをパッチごとに個別に実行する必要があるため、非常に遅く、また、パッチの重複により冗長性が多くなります。第二に、位置推定精度とコンテキストの利用の間にはトレードオフがあります。パッチが大きいほど、より多くのMax-Pooling層が必要になり、位置推定精度が低下します。一方、パッチが小さいと、ネットワークはわずかなコンテキストしか認識できません。より最近のアプローチ[11,4]では、複数の層からの特徴を考慮した分類器出力が提案されています。これにより、優れた位置推定とコンテキストの利用が同時に可能になります。
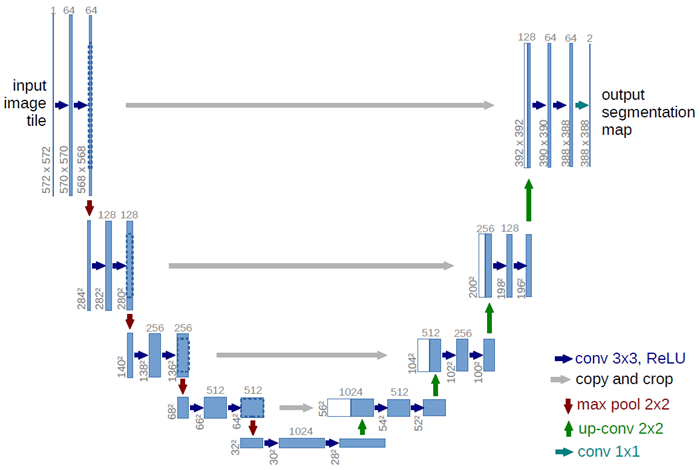
本論文では、より洗練されたアーキテクチャ、いわゆる「完全畳み込みネットワーク」[9]を基盤として構築します。このアーキテクチャを修正・拡張することで、非常に少ない訓練画像でも動作し、より高精度なセグメンテーションを生成できるようになります(図1参照)。[9]の主要なアイデアは、通常の収縮ネットワークを連続層で補完し、プーリング演算子をアップサンプリング演算子に置き換えることにあります。したがって、これらの層は出力の解像度を上げます。局所化を行うために、収縮パスからの高解像度の特徴がアップサンプリングされた出力と組み合わせます。連続畳み込み層は、この情報に基づいてより高精度な出力を組み立てることを学習することができます。

図1. U-netアーキテクチャ(最低解像度の32x32ピクセルの例)。青いボックスはそれぞれ、マルチチャンネルの特徴マップに対応しています。チャンネル数はボックスの上部に表示されています。x-yサイズはボックスの左下に表示されます。白いボックスはコピーされた特徴マップを表します。矢印は異なる操作を示しています。
私たちのアーキテクチャにおける重要な変更点の一つは、アップサンプリング部分に多数の特徴チャネルを追加したことです。これにより、ネットワークはコンテキスト情報を高解像度層に伝播できます。その結果、拡張パスは縮小パスとほぼ対称となり、U字型のアーキテクチャとなります。ネットワークには全結合層がなく、各畳み込みの有効な部分のみを使用します。つまり、セグメンテーションマップには、入力画像で完全なコンテキストが利用可能なピクセルのみが含まれます。この戦略により、オーバーラップタイル戦略(図2参照)によって任意の大きさの画像をシームレスにセグメンテーションできます。画像の境界領域のピクセルを予測するために、欠落しているコンテキストは入力画像をミラーリングすることで外挿されます。このタイリング戦略は、ネットワークを大きな画像に適用する上で重要です。そうでなければ、解像度がGPUメモリによって制限されてしまうからです。
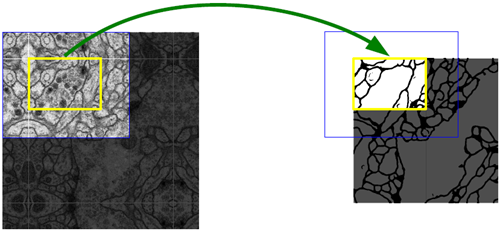
図2. 任意の大きな画像(ここではEMスタック内の神経構造のセグメンテーション)をシームレスに分割するためのオーバーラップタイル戦略。黄色の領域におけるセグメンテーションの予測には、青色の領域内の画像データを入力として必要とします。欠損した入力データは、ミラーリングによって外挿されます。
私たちのタスクでは利用可能なトレーニングデータが非常に少ないため、利用可能なトレーニング画像に弾性変形を適用することで、大幅なデータ拡張を行います。これにより、ネットワークは、注釈付き画像コーパスでこれらの変形を確認する必要なく、そのような変形に対する不変性を学習できます。これは、変形がかつて組織における最も一般的な変化であり、現実的な変形を効率的にシミュレートできるため、生物医学的セグメンテーションにおいて特に重要です。不変性学習におけるデータ拡張の価値は、Dosovitskiyら[2]によって、教師なし特徴学習の分野で示されています。
多くの細胞セグメンテーションタスクにおけるもう1つの課題は、同じクラスの接触している物体を分離することです(図3を参照)。この目的のために、接触している細胞間の分離背景ラベルが損失関数において大きな重みを得る重み付き損失の使用を提案します。
得られたネットワークは、様々な生物医学的セグメンテーション問題に適用可能です。本論文では、EMスタック(ISBI 2012で開始された継続中のコンペティション)における神経構造のセグメンテーションの結果を示し、Ciresanら[1]のネットワークを上回る性能を示しました。さらに、ISBI細胞追跡チャレンジ2015の光学顕微鏡画像における細胞セグメンテーションの結果も示します。この結果では、最も難易度の高い2つの2D透過光データセットにおいて、大きな差をつけて勝利しました。
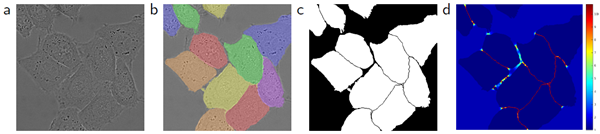
図3. DIC(微分干渉コントラスト)顕微鏡で記録されたガラス上のHeLa細胞。(a) 生画像。(b) 正解セグメンテーションのオーバーレイ。異なる色はHeLa細胞の異なるインスタンスを示す。(c) 生成されたセグメンテーションマスク(白:前景、黒:背景)。(d) ネットワークに境界ピクセルを学習させるためのピクセル単位の損失重み付けマップ。
ネットワークアーキテクチャを図1に示します。これは、縮小パス(左側)と拡張パス(右側)で構成されています。縮小パスは、畳み込みネットワークの典型的なアーキテクチャに従っています。2つの3x3畳み込み(パディングなし畳み込み)を繰り返し適用し、それぞれにReLU(Rerectified Linear Unit)とストライド2の2x2最大プーリング演算を適用してダウンサンプリングを行います。ダウンサンプリングの各ステップで、特徴チャンネル数を2倍にします。拡張パスの各ステップは、特徴マップのアップサンプリング、特徴チャンネル数を半分にする2x2畳み込み(「アップ畳み込み」)、縮小パスから切り取られた特徴マップとの連結、そして2つの3x3畳み込み(それぞれにReLUを適用)から構成されます。畳み込みのたびに境界ピクセルが失われるため、クロッピングが必要になります。最終層では、1x1畳み込みを用いて、64要素の特徴ベクトルをそれぞれ必要な数のクラスにマッピングします。ネットワークは合計23の畳み込み層を持ちます。
出力セグメンテーションマップのシームレスなタイリングを可能にするには(図2参照)、すべての2x2 Max-Pooling演算がX軸とY軸のサイズが均一なレイヤーに適用されるように入力タイルのサイズを選択することが重要です。
入力画像とそれに対応するセグメンテーションマップを用いて、Cae [6] の確率的勾配降下法を用いてネットワークを学習します。パディングなしの畳み込みのため、出力画像は入力画像よりも一定の境界幅だけ小さくなります。オーバーヘッドを最小限に抑え、GPUメモリを最大限に活用するために、大きなバッチサイズよりも大きな入力タイルを優先し、バッチを1枚の画像に縮小します。そのため、高いモーメンタム(0.99)を使用し、以前に使用した多数の学習サンプルに基づいて、現在の最適化ステップでの更新を決定します。
エネルギー関数は、最終的な特徴マップに対してピクセル単位のソフトマックス法と交差エントロピー損失関数を組み合わせることで計算されます。ソフトマックス法は\(p_k(x) = exp(a_k(x))=/\left(\sum_{k^\prime=1}^K exp(ak^\prime (x))\right)\)と定義されます。ここで、\(a_k(x)\)は、\(\Omega\subset\mathbb Z^2\)におけるピクセル位置\(x\in\Omega\)における特徴チャネルkの活性化を表します。Kはクラス数、\(p_k(x)\)は近似最大値関数です。すなわち、最大の活性化\(a_k(x)\)を持つ\(k\)については\(p_k(x)\approx 1\)、それ以外のすべての\(k\)については\(p_k(x)\approx 0\)とします。そして、クロスエントロピーは各位置において\(p_{l(x)}(x)\)の1からの偏差を次のようにペナルティとして課します。 \[ E =\sum_{x\in\Omega} w(x)\log(p_{l(x)}(x)) \tag{1} \] ここで、\(l:\Omega \rightarrow \{1,\cdots,K\}\) は各ピクセルの真のラベルであり、\(w :\Omega \rightarrow \mathbb R\) はトレーニングで一部のピクセルにさらに重要性を与えるために導入した重みマップです。
学習データセット内の特定のクラスのピクセルの頻度の違いを補正し、接触する細胞間に導入する小さな分離境界をネットワークに学習させるために、各正解セグメンテーションの重みマップを事前に計算します(図3cおよびdを参照)。
分離境界は形態学的演算を用いて計算される。重みマップは次のように計算されます。 \[ w(x) = w_c(x) + w_0\cdot exp\left(-\frac{(d_1(x)+d_2(x))^2}{2\sigma^2}\right) \tag{2} \] ここで、\(w_c :\Omega\rightarrow \mathbb R\) はクラス頻度のバランスをとるための重みマップ、\(d_1:\Omega\rightarrow \mathbb R\) は最も近いセルの境界までの距離、\(d_2 :\Omega\rightarrow \mathbb R\) は2番目に近いセルの境界までの距離を表します。実験では、\(w_0 = 10\) および \(\sigma \approx 5\) ピクセルに設定しました。
多数の畳み込み層とネットワークを介した多様な経路を持つ深層ネットワークでは、重みの適切な初期化が極めて重要です。適切に初期化しないと、ネットワークの一部が過剰な活性化を引き起こし、他の部分は全く寄与しない可能性があります。理想的には、ネットワーク内の各特徴マップがほぼ単位分散を持つように初期重みを調整する必要があります。私たちのアーキテクチャ(畳み込み層とReLU層を交互に配置)を持つネットワークでは、標準偏差が\(\sqrt{2/N}\)のガウス分布から初期重みを取得することでこれを実現できます。ここで、\(N\)は1つのニューロンの入力ノード数を表します[5]。例えば、3x3の畳み込み層と前の層の64個の特徴チャネルの場合、\(N = 9\cdot 64 = 576\)となります。
データ拡張は、利用可能なトレーニングサンプルが少ない場合に、ネットワークに必要な不変性と堅牢性を学習させるために不可欠です。顕微鏡画像の場合、主にシフトと回転に対する不変性、そして変形とグレー値の変化に対する堅牢性が必要です。特に、トレーニングサンプルのランダムな弾性変形は、非常に少ない注釈付き画像でセグメンテーションネットワークをトレーニングするための鍵となる概念です。粗い3×3グリッド上のランダム変位ベクトルを用いて、滑らかな変形を生成します。変位は、標準偏差10ピクセルのガウス分布からサンプリングされます。その後、ピクセルごとの変位は双3次補間を用いて計算されます。収縮パスの最後にあるドロップアウト層は、さらに暗黙的なデータ拡張を実行します。
u-netを3つの異なるセグメンテーションタスクに適用する例を示します。最初のタスクは、電子顕微鏡記録における神経構造のセグメンテーションです。データセットの例と得られたセグメンテーションを図2に示します。完全な結果は補足資料として提供する。データセットは、ISBI 2012で開始され、現在も新しい投稿を募集しているEMセグメンテーションチャレンジ[14]によって提供されました。トレーニングデータは、ショウジョウバエ1齢幼虫腹側神経索(VNC)の連続切片透過型電子顕微鏡画像から得られた30枚の画像(512×512ピクセル)です。各画像には、細胞(白)と膜(黒)の対応する完全に注釈付けされた正解セグメンテーションマップが付属しています。テストセットは公開されていますが、セグメンテーションマップは非公開です。評価版を入手するには、予測された膜確率マップを主催者に送付します。評価は、マップを10の異なるレベルで閾値処理し、「ワーピング誤差」、「ランダム誤差」、および「ピクセル誤差」を計算することによって行われます[14]。
u-net(入力データの7つの回転バージョンを平均)は、追加の前処理や後処理を行わなくても、ワーピング誤差0.0003529(新しい最高スコア、表1を参照)とrand誤差0.0382を達成しました。
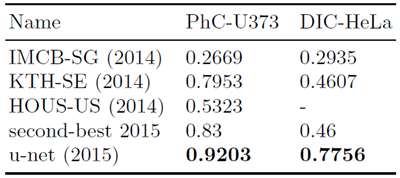
表1. EMセグメンテーションチャレンジ[14](2015年3月6日)におけるワーピングエラー順のランキング。
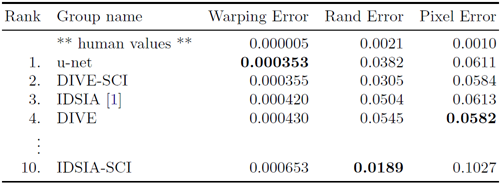
これは、Ciresanら[1]によるスライディングウィンドウ畳み込みネットワークの結果よりも大幅に優れています。Ciresanらの最高の提出物は、ワーピング誤差が0.000420、rand誤差が0.0504でした。rand誤差に関して、このデータセットでより優れたパフォーマンスを発揮するアルゴリズムは、Ciresanら[1]の確率マップにデータセット固有の高度な後処理手法1を適用したアルゴリズムのみです。
1 このアルゴリズムの著者は、この結果を達成するために78通りの異なる解法を提示しました。
我々はまた、u-netを光学顕微鏡画像における細胞セグメンテーションタスクに適用した。このセグメンテーションタスクは、ISBI細胞追跡チャレンジ2014および2015 [10,13]の一部である。最初のデータセット PhC-U373"2には、位相差顕微鏡で記録されたポリアクリルイミド基板上の神経膠腫・星状細胞腫U373細胞が含まれている(図4a、bおよび補足資料を参照)。このデータセットには、部分的に注釈が付けられた35枚のトレーニング画像が含まれている。ここでは平均IOU(intersection over union)が92%となり、2番目に優れたアルゴリズムの83%を大幅に上回った(表2を参照)。 2番目のデータセット「DIC-HeLa」3は、微分干渉(DIC)顕微鏡で記録されたガラス上のHeLa細胞です(図3、図4c、d、および補足資料を参照)。このデータセットには、部分的に注釈が付けられた20枚の学習画像が含まれています。ここでは平均IOUが77.5%に達し、2番目に優れたアルゴリズムの46%を大幅に上回っています。
2
データセット提供:サンジェイ・クマール博士(カリフォルニア大学バークレー校バイオエンジニアリング学部、カリフォルニア州バークレー(米国))
3
データセットはゲルト・ファン・カペレン・エラスムス医療センター博士から提供されました。ロッテルダム。
オランダ
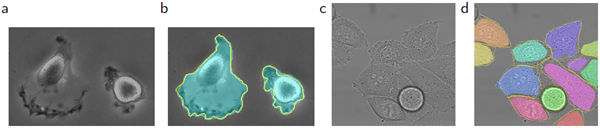
図4. ISBI細胞追跡チャレンジの結果。(a) PhC-U373データセットの入力画像の一部。(b) 手動で作成した正解(黄色の枠線)を使用したセグメンテーション結果(シアンマスク)。(c) \DIC-HeLaデータセットの入力画像。(d) 手動で作成した正解(黄色の枠線)を使用したセグメンテーション結果(ランダムカラーマスク)。
表 2. ISBI 細胞追跡チャレンジ 2015 におけるセグメンテーション結果 (IOU)。
u-netアーキテクチャは、非常に多様な生物医学セグメンテーションアプリケーションにおいて非常に優れたパフォーマンスを発揮します。弾性変形によるデータ拡張により、必要な注釈付き画像はごくわずかで、NVidia Titan GPU (6 GB) でわずか10時間という非常に合理的な学習時間を実現しています。私たちは、Cae[6]ベースの完全な実装と学習済みネットワーク4を提供します。u-netアーキテクチャは、より多くのタスクに容易に適用できると確信しています。
この研究は、ドイツ連邦政府および州政府のエクセレンス・イニシアチブ(EXC 294)およびBMBF(Fkz 0316185B)の支援を受けて実施されました。